1、芥菜A亚基因组起源问题
芥菜的基因组是异源四倍体(AABB),在“禹氏三角”中由白菜(AA),黑芥(BB)杂交后加倍形成,在演化过程中变异类型非常丰富。问题是油用芥菜的AA和菜用芥菜的AA是来自同一个亚种,还是来自多个亚种呢,这个问题就是A亚基因组的起源问题。
如上图,a中对芥菜A、白菜A、甘蓝型油菜A进行共线性分析,可以发现其是高度共线的。
我们对10个菜用的芥菜、7个油用的芥菜,5个甘蓝型油菜基因组、27个白菜基因组(多亚种)进行了重测序分析,并绘制如上图b中的进化树。从b图中可以看到芥菜全部聚在一起,没有出现分散的情况,说明芥菜中A的基因组是来源于同一个亚种,属于单系起源。
C图中对同源物种和芥菜进行了进化树构建,并计算了芥菜分化的具体时间为3-5万年。
除了从群体的角度研究了芥菜亚基因组A起源问题,还从PCA聚类和Fixed SNP角度验正了单系起源的结论。
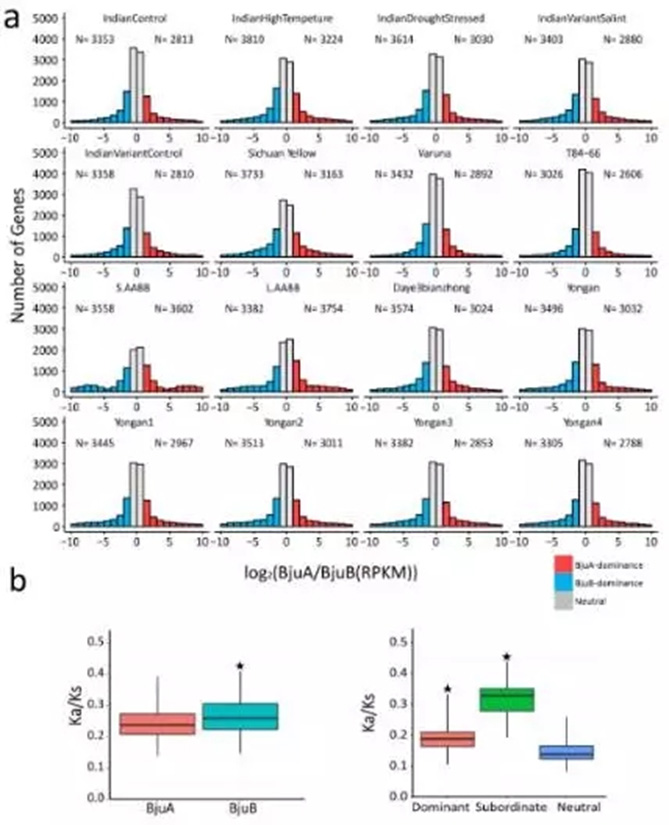
2、基因表达的dominance现象
由于芥菜基因组是异源四倍体,也就是说基因组中存在两套非常相似的亚基因组,那么在基因表达的过程中,位于两套亚基因组上的等位基因的表达模式是怎么样的呢,是一起表达,是相互抑制,还是一方占主导?
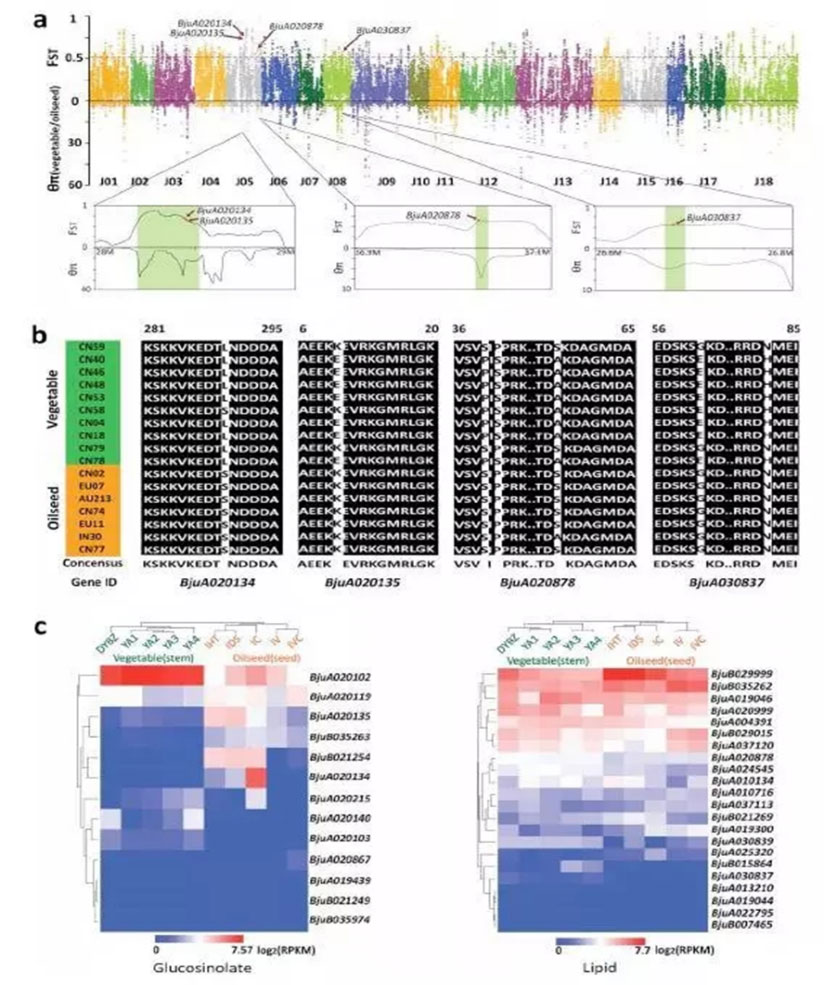
3、油用芥菜和菜用芥菜的选择与分化
通过菜用和油用芥菜群体进行选择清除分析,发现dominance的基因被筛选出来的比例较高,同时结合转录组数据,这部分基因在油用和菜用两个群体中差异表达。同时通过上面的分析发现与硫苷,脂类代谢显著相关并且存在dominance的基因组,这些基因在油用菜用群体中有各自独特基因分型。

 技术优势
技术优势 技术路线
技术路线 分析内容
分析内容 样本类型
样本类型 案例展示
案例展示 研究背景
研究背景 研究结果
研究结果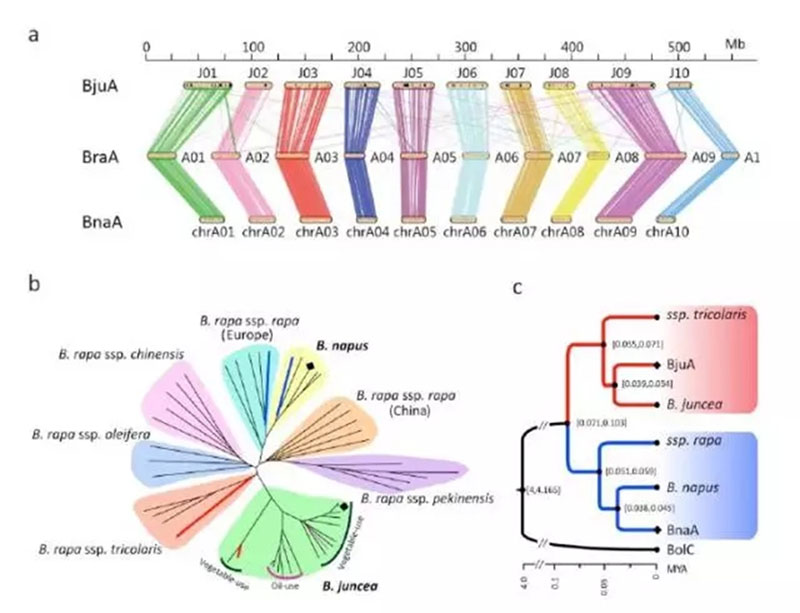
 参考文献
参考文献 结果展示
结果展示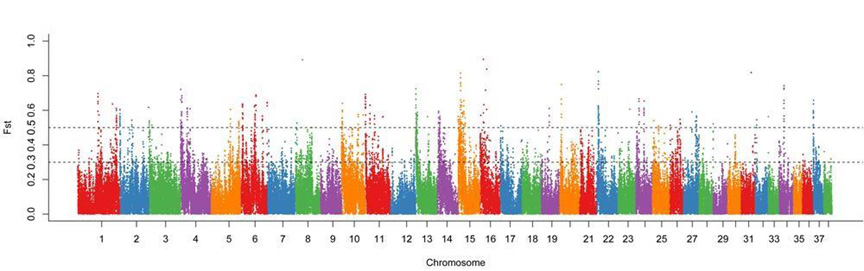
 常见问题
常见问题