转录组数据数据这么多,怎么办?怎么看?和我的研究有什么关系?
如何分析?如何从大海中捞到那根针呢?
别急,小编抛砖引玉啦~
Part1 测序数据质控评估
1.1测序数据质控评估
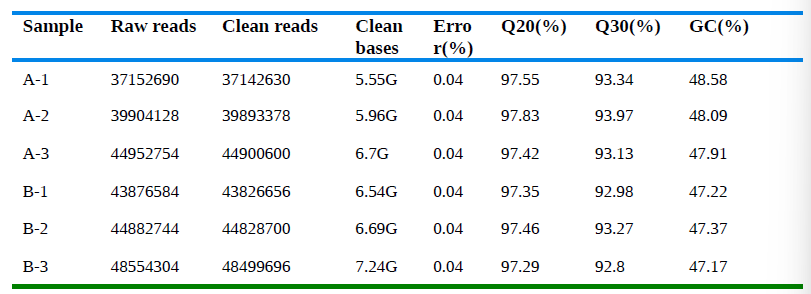
测序错误率与碱基质量有关,受测序仪本身、测序试剂、样本等多个因素的共同影响。如何评估呢?可从以下几个维度进行测序数据质量评估:1)用于后续分析的测序数据量(clean bases),如果数据量>6G,那么ok,基本上满足分析要求;如果需要更多数据量进行可变剪切或融合基因分析,没问题,加测即可;2)碱基质量值(Q20/Q30)百分比,Q20/Q30>85%-90% 说明测序质量过关。如果测序数据量和碱基质量值符合质控要求,就可以放心的往下看了呢~
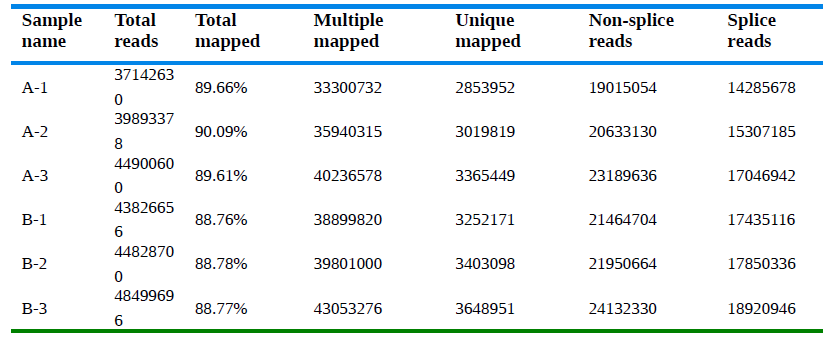
1.2参考基因组比对
如果物种基因组注释比较好(如人/大鼠/小鼠等),参考基因组比对率(total mapped)一般要求是>85%(如 >85%的基因组比对率表示测序到的reads 85%以上属于该物种,说明测序样本的物种信息无误)。但如果是非常规物种或基因组注释不那么完整,那么基因组比对率要求就会适当低一些呢(>60-85%,各位看官根据实际情况进行综合判断哈)。
1.3样本生物学重复评估
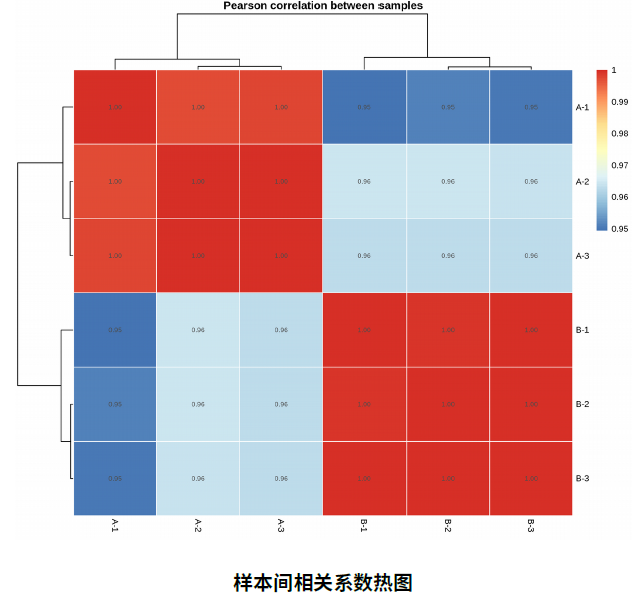
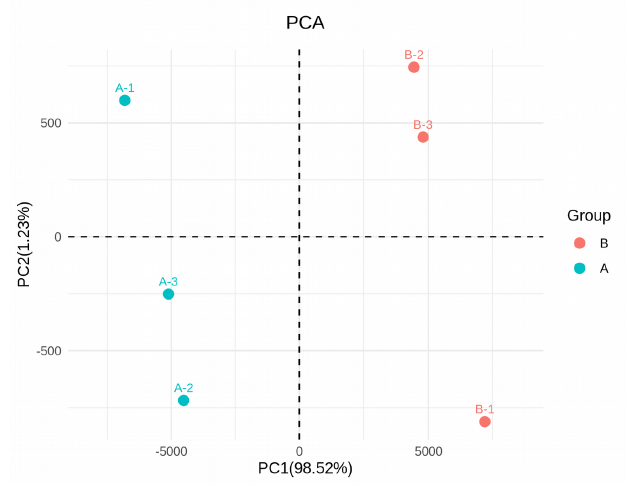
假如实验设计(比较组)是 Case组 vs Control 组 、敲除/过表达组 vs 对照组等,如何看2个比较组在组学(mRNA)层面上是否有组间差异呢?样本间相关性系数分析和PCA可以提供答案。
(1)相关性系数越接近1,表明样本之间表达模式的相似度越高;对于同一组的生物学重复样本来说(如A1/A2/A3/A4/A5…),相关性系数越接近说明同组内的生物学重复越好;不同组别(A组和B组)之间的相关性系数相差越大越好,说明不同组别之间的差别越大,组间的差异基因可能越多;
(2)同样,同一组的样本越接近说明组内生物学重复越好;不同组别的样本在PCA中距离越远越好,说明组间差异越大。当然除了样本间相关性系数分析和PCA分析外,还有其他分析方法,在此先不赘述了呢~
Part2筛选差异/候选基因及结果可视化
2.1筛选差异/候选基因
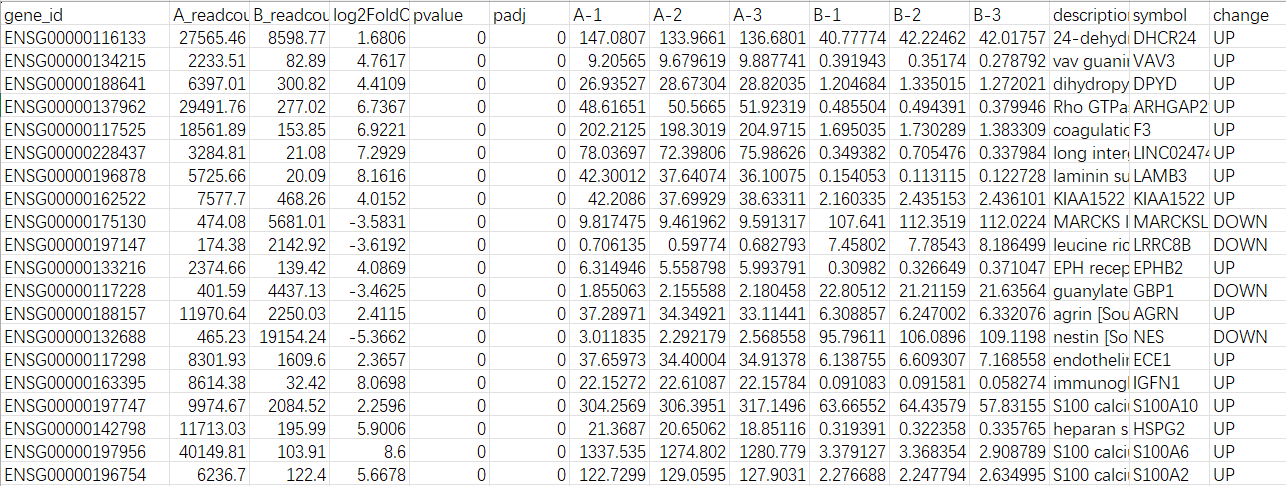
默认将padj小于0.05,|log2(foldchange)| 大于1作为筛选差异基因的标准(见下表),可发现哪些基因在2组中发生了显著性变化(显著上调或者显著下调表达),这些显著变化基因极有可能和处理组/疾病组等显著相关,可作为后续研究的候选基因。
如果筛选出来的差异基因可能比较多怎么办呢?通过差异倍数(FoldChange)、padj/p(越小越好)、基因在各个样本的中表达情况(FPKM)、基因功能注释等这些因素进行进一步筛选,以进一步缩小候选基因范围。如果差异基因较少,反其道行之,放宽筛选标准,使更多的基因出现。当然,实际筛选过程中,依据实际项目情况进行个性化调整~
2.2 差异/候选基因可视化展示
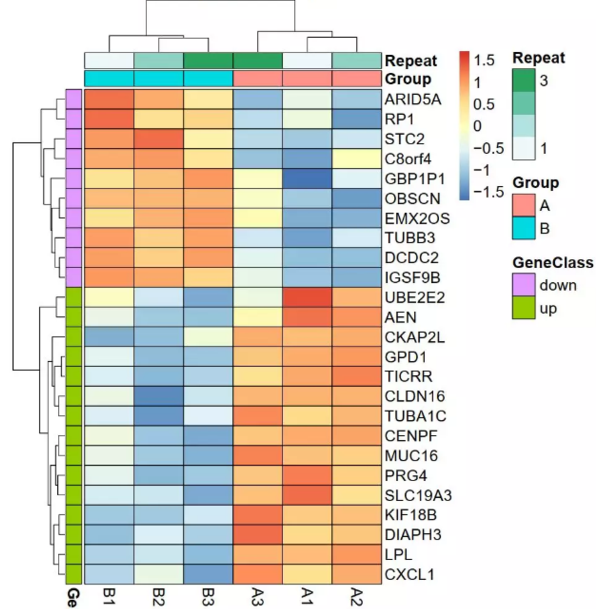
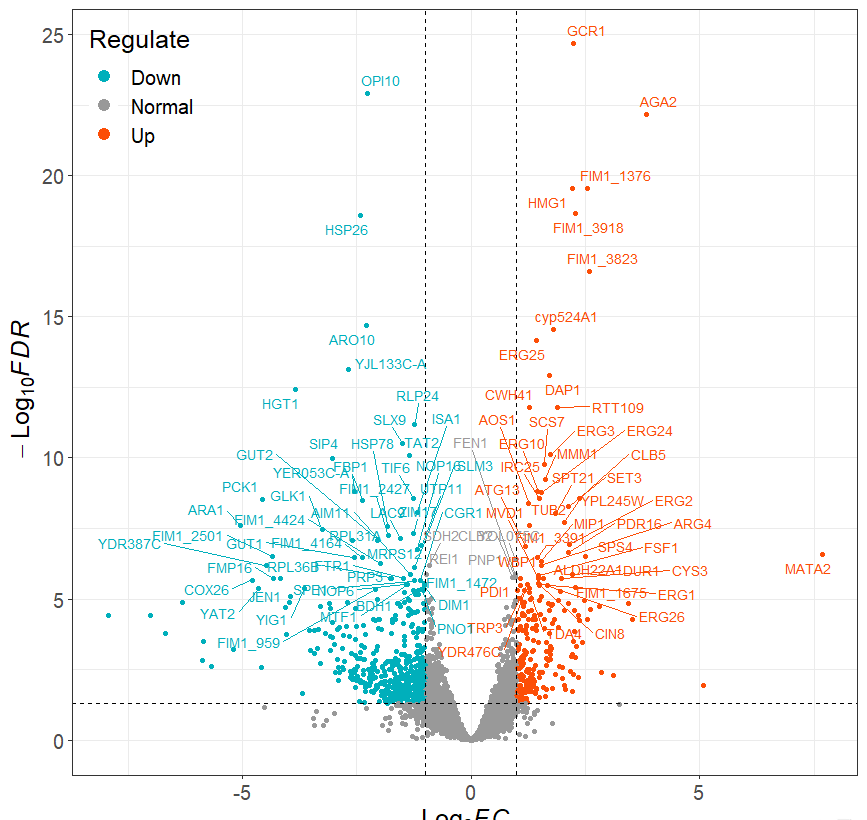
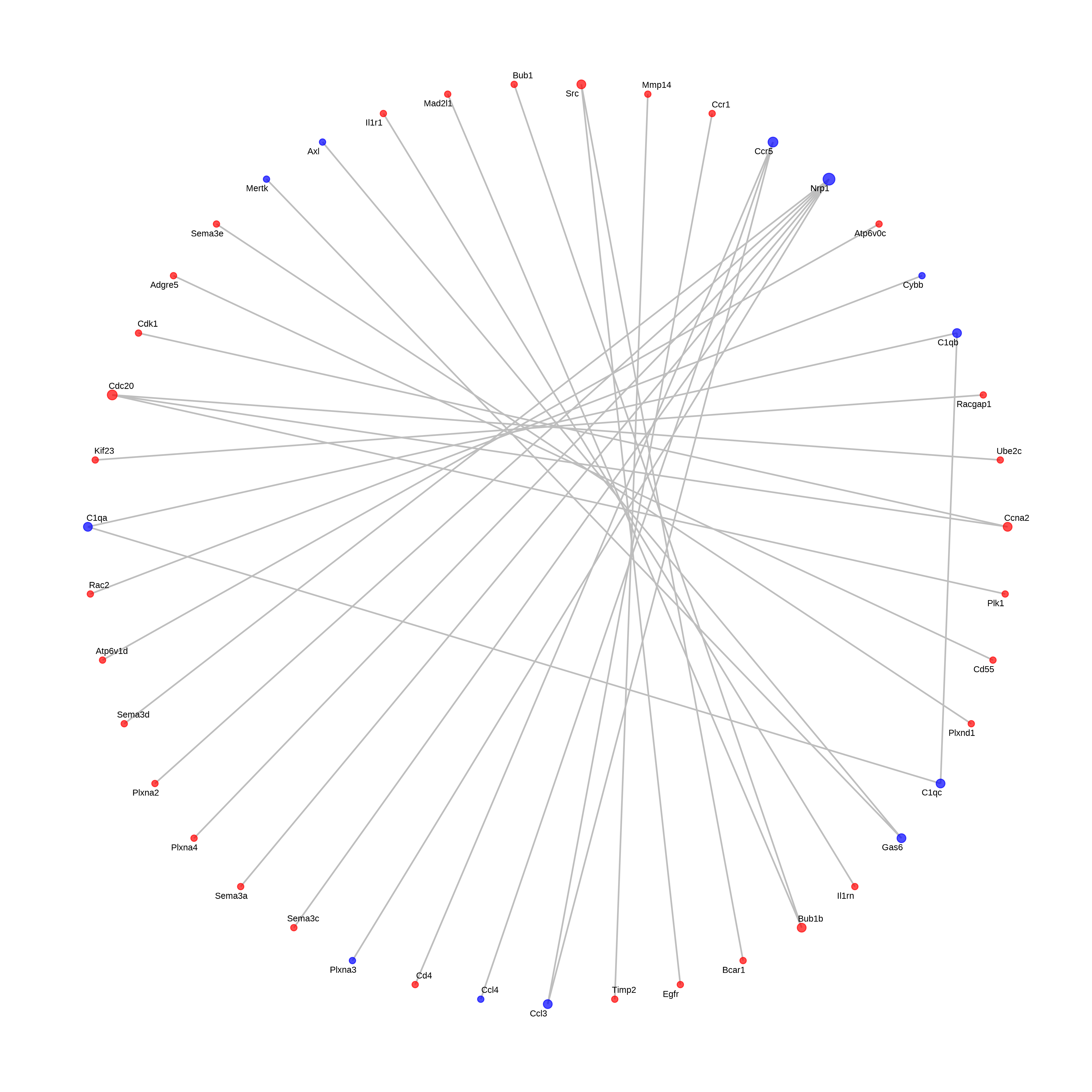
筛选出差异/候选基因后(建议10-100个基因内),如果对这些基因进行可视化展示呢?热图、火山图、PPI网络图来啦~
热图可以清楚地展示基因在各个分组/样本中的表达情况(颜色越红表达越高);火山图可以特异性展示差异/候选基因的差异倍数和显著性情况;PPI网路图可以帮助展示这些基因之间的相互作用(基因A与其他基因的连线越多,说明与其互作的基因数越多,基因A调控/受调控的基因越多,基因A的作用可能越重要)。
Part3功能富集分析及结果可视化
3.1 差异基因功能富集分析
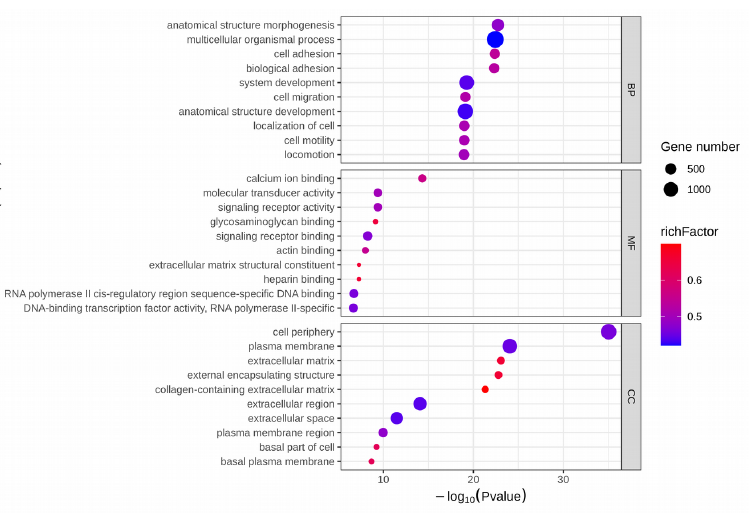
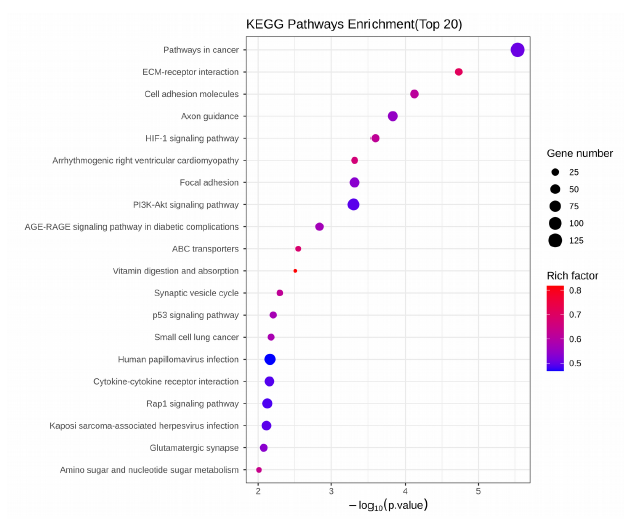
差异基因的GO/KEGG富集散点图是富集分析结果的一种可视化展示方式。默认挑选富集显著的 Top20 GO term/pathway进行展示,此图中通过Rich factor、P value 和Gene number (富集到此通路上的基因个数)来衡量其富集程度。当然,如果有特别关注/感兴趣的GO term/pathway,也可以针对目标term 进行个性化调整,以实现对特定功能类别的聚焦。
3.2 GSEA富集分析
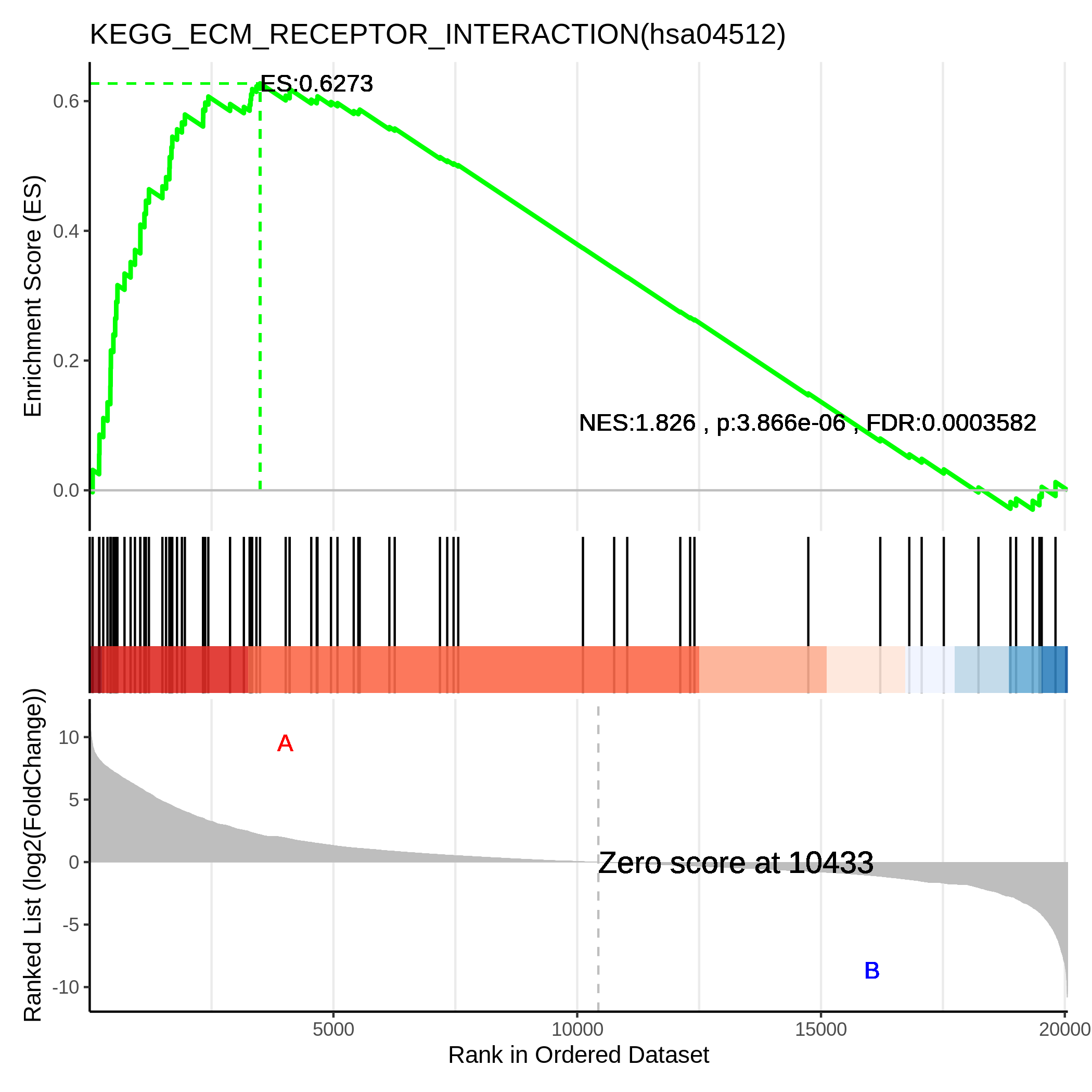
GSEA 不关注某几个表达发生显著改变的基因,而是整个表达数据在特定功能基因集中的表达一致性,以此来解读数据中蕴含的生物学信息。因此GSEA可以避免差异表达分析中阈值筛选带来的问题。如下图,此图中主要通过NES、P/FDR (显著性,越小越显著)来衡量其富集程度。NES正值表示在左侧(A组)组别中显著性富集,说明该term 在A组中处于激活状态,反之在B组中处于抑制状态;NES负值表示在右侧(B组)组别中显著性富集,说明该term 在B组中处于激活状态。如果有特别关注/感兴趣的GO term/pathway,可以单独展示其富集情况哈~
Part4个性化分析
4.1趋势分析
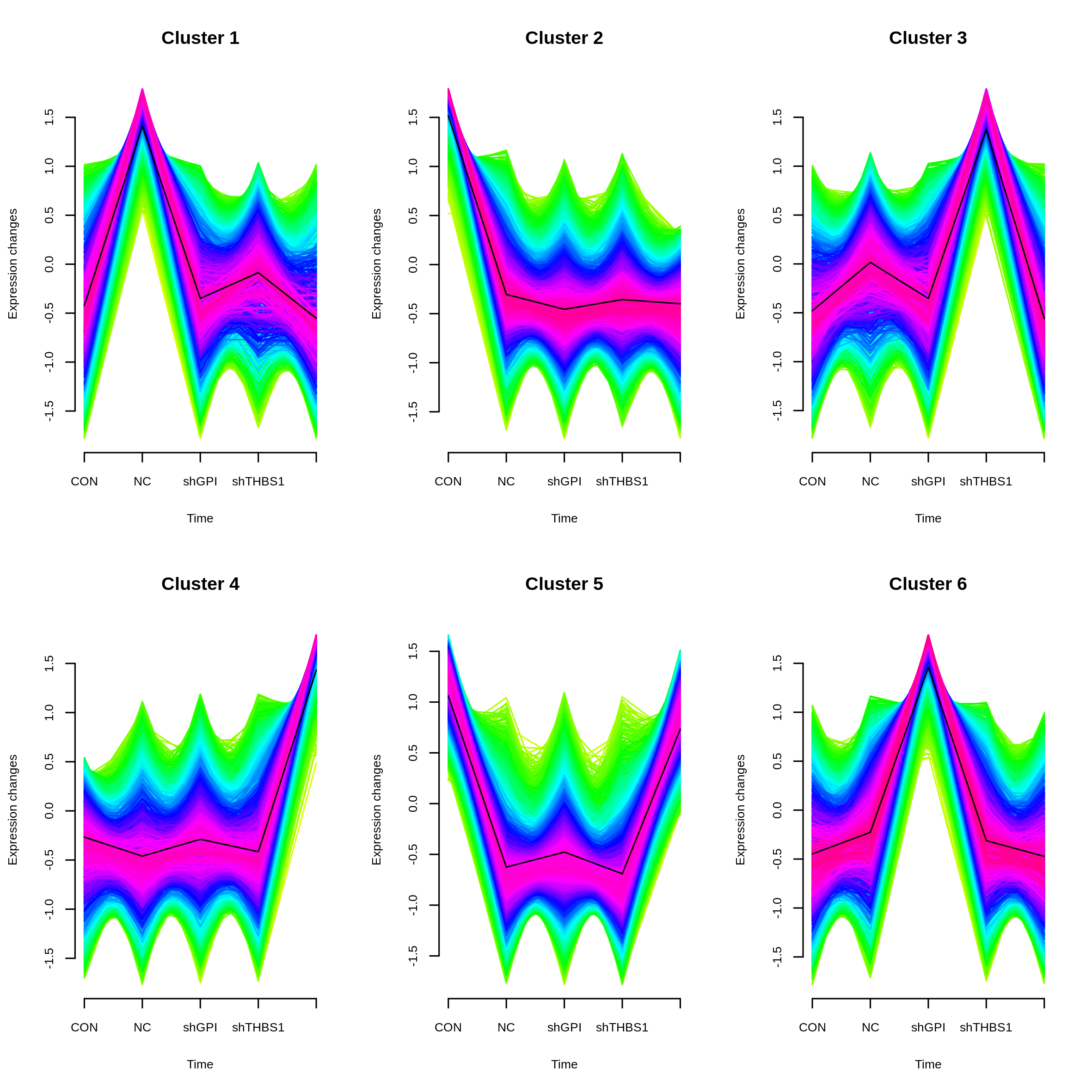
趋势分析为梯度类文章的核心分析点,如实验设计(3-5组)涉及梯度处理(时间变化、药物浓度/剂量变化、疾病程度、生长周期等),利用趋势分析/时间序列分析将表达模式相似的基因进行归类,从而找到实验变化过程中最具有代表性的基因集及对应的趋势特征(如cluster),揭示在变化过程中所特有的规律。筛选到与预期表达趋势符合的cluster后,可以对特定cluster中基因进行针对性分析(如表达分析、互作分析、功能分析等)。
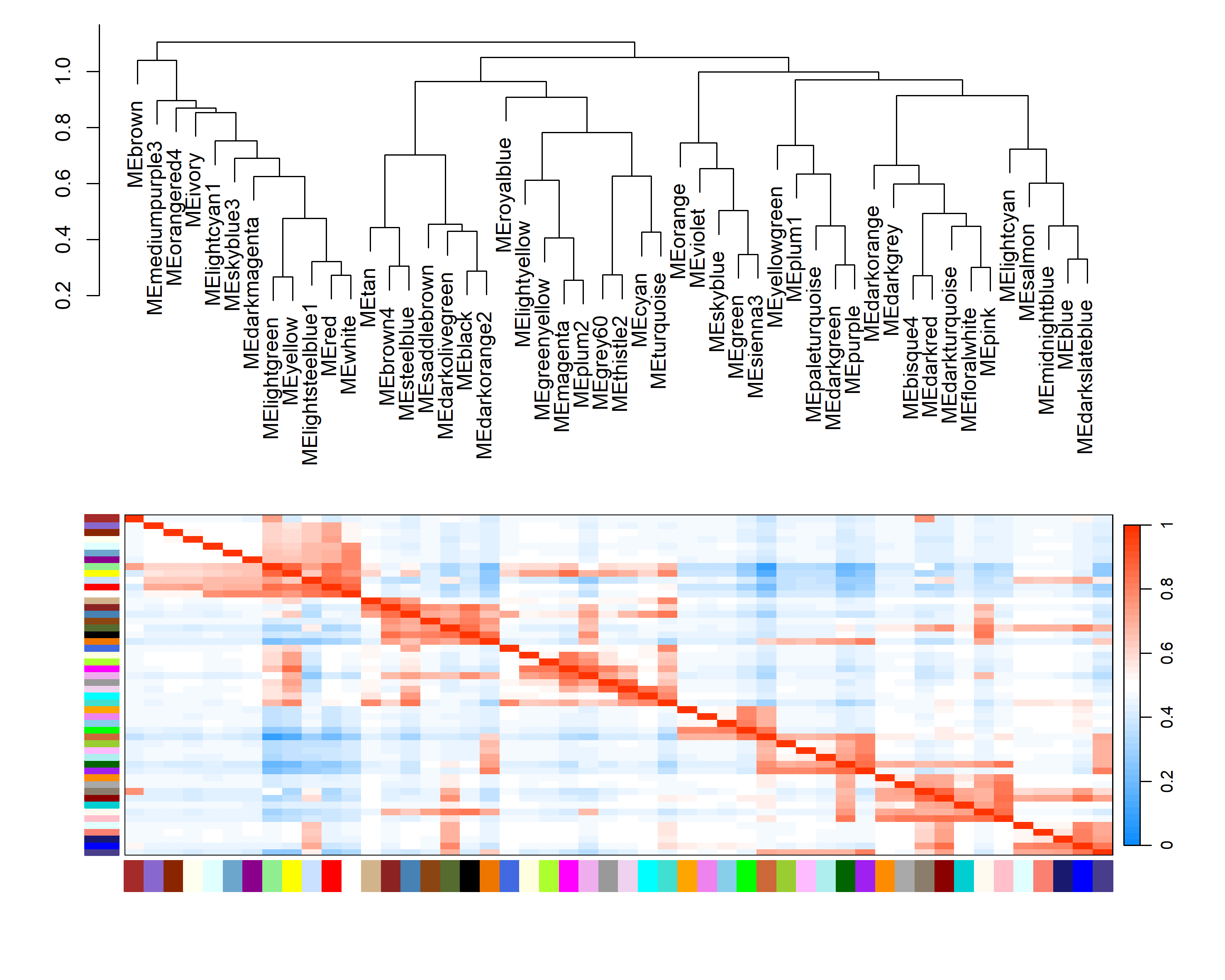
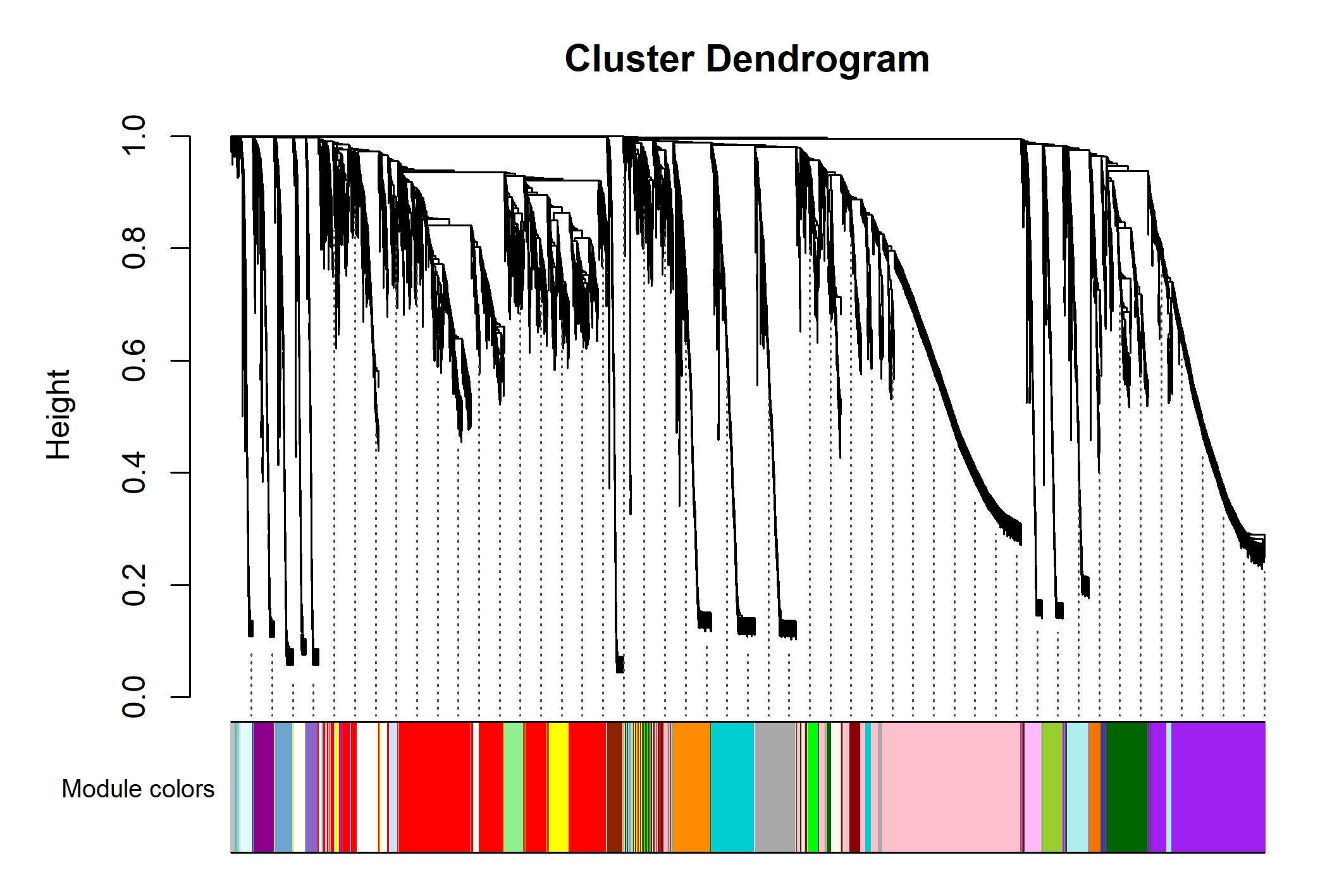
4.2 WGCNA分析
WGCNA(Weighted Gene Co-expression Network analysis),即加权基因共表达网络分析。WGCNA相当于对多个复杂分组进行分析,用于找寻不同分组/表型的特征基因模块,从而进行下一步分析(如可以对模块内基因进行GO/KEGG富集、PPI等等)。
Tips:课题设计咨询、详细结果解释/沟通、个性化分析需求等,欢迎随时联系我们的科研顾问或销售小伙伴哈~