
癌细胞还能这样杀?!“i-CRISPR”策略——打断癌细胞特有的DNA,却阻止其修复
目前,手术、化疗和放疗是治疗癌症的主要方式,因个体化的差异导致很大一部分患者不能完全治愈[1,2]。仅通过手术通常无法清除所有的癌细胞,而其他放疗、化疗等策略在杀死癌细胞的同时,往往会对正常组织造成严重的损害[3,4]。因此,开发一种既能特异性杀伤癌细胞又不会对正常细胞造成明显损伤的策略,对癌症治疗具有重要的临床意义。
不可修复的DNA损伤会导致细胞死亡,癌症的放射治疗正是利用了该原理,通过电离辐射对细胞诱导不可修复的DNA损伤,不过放疗造成的DNA双链断裂(DSBs)是随机的,不具有特异性,难以避免对正常细胞的损伤[5,6]。
随着科学技术的不断发展,新兴的CRISPR-Cas9基因编辑技术可以实现在特定位点精准创建DSBs[7-9]。在高等真核生物中,DSBs会通过非同源末端连接(nonhomologous end joining, NHEJ)和同源重组(homologous recombination, HR)修复途径对其进行修复[10,11]。如果在抑制上述DNA修复通路的同时,利用CRISPR-Cas9基因编辑技术仅对癌细胞特有的DNA进行精准切割,是否就能特异杀伤癌细胞了呢?这是海军军医大学的研究者们创造性地提出的一个癌细胞特异性杀伤的构想。并且该构想被实验证实,相关结果近期以“i-CRISPR:a personalized cancer therapy strategy through cutting cancer-specific mutations”为题,在线发表在国际权威期刊Molecular Cancer (IF=41.444) 上。海军军医大学王越,杨彦勇,高福,韩超峰教授为文章的共同通讯作者,蒋俊锋,陈媛媛和张莉为本文的并列第一作者。

研究提出的“i-CRISPR”策略,就是根据癌细胞的DNA序列,定制且导入仅能切割癌细胞DNA的CRISPR-Cas9系统——“癌细胞CRISPR剪刀”,造成癌细胞中DNA断裂;同时添加DNA损伤修复抑制剂(DNA damage repair inhibitor, DSBRi),使癌细胞中断裂的DNA无法修复,导致细胞死亡。DNA损伤修复抑制剂就是“i-CIRSPR”策略中的“i”,切割癌细胞DNA的“癌细胞CRISPR剪刀”就是“i-CIRSPR”策略中的“CRISPR”。 由于正常细胞中不含有可被“癌细胞CRISPR剪刀”切割的位点,因此其DNA不会被切割,便不会受到严重损伤。如此,这种“i-CRISPR”策略有望实现既能特异性杀伤癌细胞又不会对正常细胞造成明显损伤的效果,该策略为肿瘤的精准治疗提供了新的治疗思路。
结果
01“i-CRISPR”个性化癌症治疗策略的设计流程
Step 1
对患者的肿瘤样本进行DNA测序,癌细胞与正常细胞比较筛选独特的DNA突变位点。
Step 2
设计特异性靶向癌细胞DNA突变位点的gRNA 用于开发CRISPR-Cas9基因编辑系统。
Step 3
导入CRISPR-Cas9基因编辑系统,同时加入DSBRi于细胞中,实现对癌细胞的特异性杀伤(图1)。

图1. “i-CRISPR”个性化癌症治疗策略流程图
02“i - CRISPR”策略通过诱导相应突变位点的DSBs杀死癌细胞
为了验证“i - CRISPR”策略,研究者首先通过全基因组测序分析了人肝癌细胞系HepG2的DNA序列,从1000多个潜在位点选取了8个候选靶点并设计相应的gRNA-Cas9,通过腺病毒载体导入HepG2细胞。体外实验验证了在CRISPR基因编辑系统导入后,HepG2细胞的编辑位点发生了DSBs事件,且DSBs标志物γH2AX表达显著上升,然而经过相同处理的Huh7细胞(无CRISPR基因编辑靶点)有较少的DSBs事件发生。
为了促进DSBs诱导的细胞死亡,研究人员使用靶向NHEJ(DNA-PKcs抑制剂NU7441)和HR(ATM抑制剂KU55933)修复的抑制剂[12,13]处理细胞,DSBs修复被显著抑制,且 NU7441 + Ku55933联合处理HepG2细胞中观察到更多未被修复的DSBs。此外,该策略采用gRNA-Cas9慢病毒系统导入前列腺癌细胞系DU145中,在更换了DNA损伤修复的抑制剂后也同样获得了促进细胞凋亡的效果,且不具有耐药性。但对正常293T细胞(无CRISPR基因编辑靶点)的活力无抑制作用。
随后,作者建立的类器官模型和人源肿瘤组织异种移植模型 (PDX)研究证明 “i - CRISPR”策略有效抑制肿瘤生长,小鼠体重、血生化以及血常规等数据也能够证明该策略具有良好的生物安全性(图2)。体内外实验结果证明,利用CRISPR技术对突变位点特异性基因编辑并结合DNA修复抑制剂可显著抑制肿瘤生长,该策略在肿瘤治疗中具有潜在的应用价值。
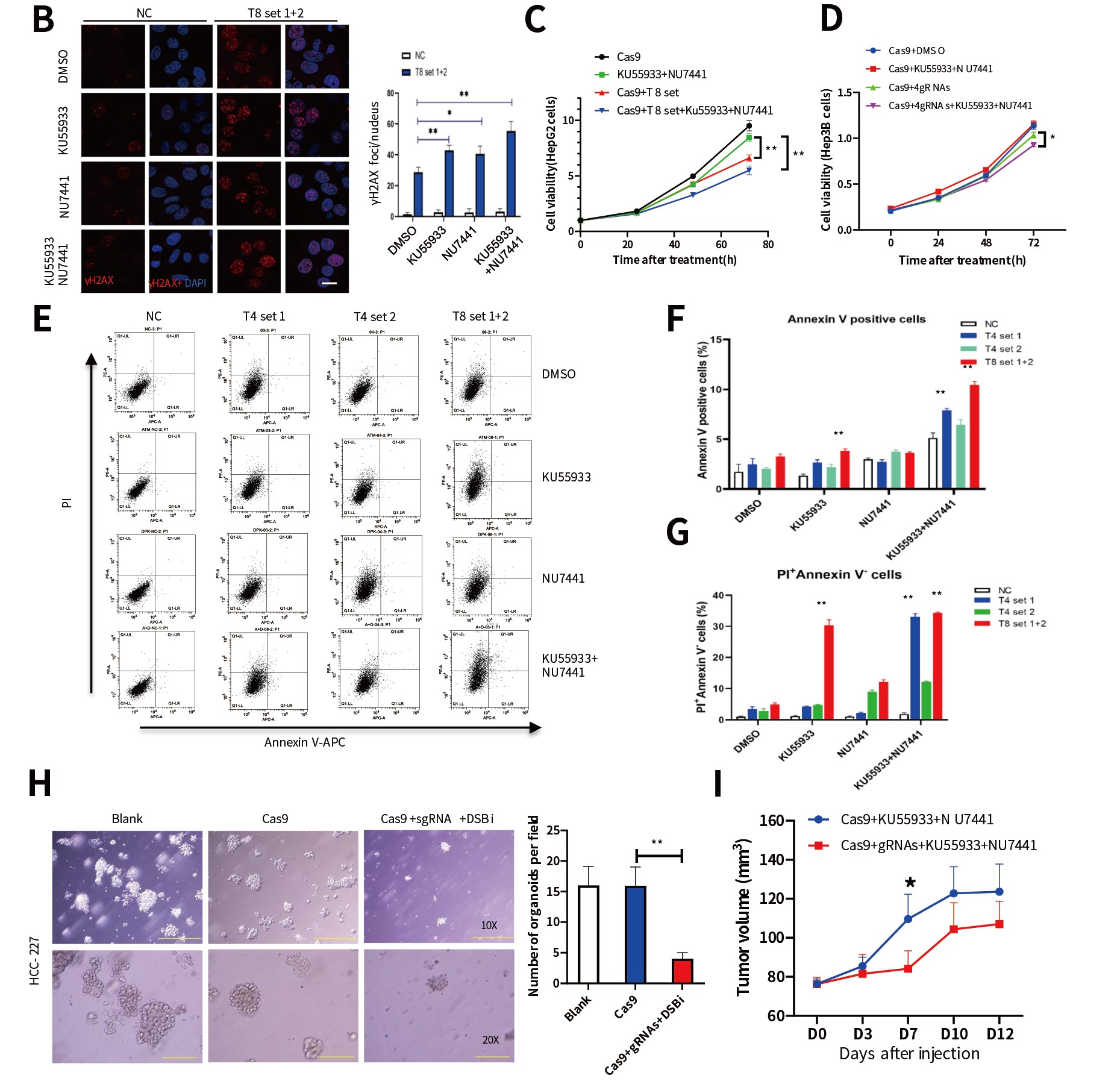
图2. “i-CRISPR”策略特异有效杀伤癌细胞
03“i - CRISPR”策略杀伤细胞的分子机制
研究者利用串联质谱对3组HepG2细胞进行定量磷酸化蛋白质组学分析来探索“i - CRISPR”策略对肿瘤细胞的杀伤机制。结果显示,在C-Cut组和NC组之间存在少量差异磷酸化蛋白,而在C-Cut-2i组中具有独特且差异较大的磷酸化蛋白主要位于细胞核中。GO分析结果显示,C-Cut组中改变的DNA损伤相关磷酸化蛋白水平增加并富集在染色质组织和DNA损伤相关通路;C-Cut-2i组的差异磷酸化蛋白富集在自噬、铁死亡、凋亡、坏死性凋亡等各种细胞死亡相关通路,尤其是自噬。因此,作者认为自噬的激活在增加C-CUT-2i组细胞死亡的分子机制中发挥了关键作用。
通过全基因组甲基化测序(WGBS)结果证明gRNA-Cas9和DSBRis策略并没有明显改变细胞系中DNA甲基化相关的表观遗传调控。但相较于对照细胞却存在一些差异甲基化区域(DMRs),且DMRs定位基因富集在细胞死亡、程序性细胞死亡和染色体组织以及凋亡通路中。
癌症治疗面临的另一个难题是不断突变的癌细胞的进化。研究者对DU145的三个单克隆株A6(培养约60代)、B12和B13(培养约80代)分别进行全基因组测序,发现单克隆株在进化过程中产生新的突变,呈现异质性。但仅有少部分是其特有的(A6: 17.11%;B12: 32.10%;B13: 38.72%),大多数原始突变得以保留。此外,本研究的DU145数据与公开的DU145突变数据进行对比也证明超过一半的原始突变仍然保留(图3)。

图3. “i-CRISPR”策略杀伤细胞的分子机制
结论
研究表明:“i - CRISPR”策略可以实现对癌细胞的特异性杀伤,而对正常细胞不造成明显损伤,并且,该策略在解决当前癌症治疗所面临的癌症进化问题上也有很大的优势。随着测序和克隆进化分析等生物信息学技术的发展,将从患者身上识别出所有癌细胞普遍存在的原始突变基因,解决癌症异质性带来的问题。通过CRISPR基因编辑系统结合DNA损伤修复抑制剂诱导肿瘤细胞特异性DSBs,加速细胞死亡,可作为肿瘤精准治疗的一种新策略,为个体化肿瘤治疗提供新思路。
参考文献:
1. Giordano SH, Elias AD, Gradishar WJ. NCCN guidelines updates: breast cancer. J Natl Compr Cancer Netw. 2018;16:605–10.
2. Noy BM, Rich BJ, Llorente R, Kwon D, Abramowitz M, Mahal B, et al. Levels of evidence for radiation therapy recommendations in the National Comprehensive Cancer Network (NCCN) clinical guidelines; 2021.
3. Nardon C. Anti-cancer chemotherapeutics in target therapy: from advanced preclinical studies on promising au(III) peptidomimetics toward the design of new receptor-recognizable metal-based agents; 2013.
4. Chang J, Mehran R, Feng L, Verma V, Liao Z, Welsh J, et al. Stereotactic ablative radiotherapy for operable stage I non-small-cell lung cancer (revised STARS): long-term results of a single-arm, prospective trial with prespecified comparison to surgery. Lancet Oncol.2021;22(10):1448–57.
5. Lord C, Ashworth A. The DNA damage response and cancer therapy. Nature. 2012;481:287–94.
6. Olivieri M, Cho T, Álvarez-Quilón A, Li K, Schellenberg M, Zimmermann M, et al. A genetic map of the response to DNA damage in human cells. Cell. 2020;182:481–496.e421.
7. Jiang J, Zhang L, Zhou X, Chen X, Huang G, Li F, et al. Induction of site-specific chromosomal translocations in embryonic stem cells by CRISPR/Cas9. Sci Rep. 2016;6:21918.
8. Bailey S, Maus M. Gene editing for immune cell therapies. Nat Biotechnol. 2019;37:1425–34.
9. Frangoul H, Altshuler D, Cappellini M, Chen Y, Domm J, Eustace B, et al. CRISPR-Cas9 gene editing for sickle cell disease and β-thalassemia. N Engl J Med. 2021;384:252–60.
10. Becker J, Clifford G, Bonnet C, Groth A, Wilson M, Chapman J. BARD1 reads H2A lysine 15 ubiquitination to direct homologous recombination. Nature.2021;596(7872):433–7.
11. Scully R, Panday A, Elango R, Willis N. DNA double-strand break repairpathway choice in somatic mammalian cells. Nat Rev Mol Cell Biol.2019;20:698–714.
12. Canny M, Moatti N, Wan L, Fradet-Turcotte A, Krasner D, Mateos-Gomez P, et al. Inhibition of 53BP1 favors homology-dependent DNA repair and increases CRISPR-Cas9 genome-editing efficiency. Nat Biotechnol. 2018;36:95–102.
13. Sultana R, Abdel-Fatah T, Abbotts R, Hawkes C, Albarakati N, Seedhouse C, et al. Targeting XRCC1 deficiency in breast cancer for personalized therapy. Cancer Res. 2013;73:1621–34.

扫一扫,反馈当前页面
和元生物